| |
| Med Sci (Paris). 2009 November; 25(11): 885–887. Published online 2009 November 15. doi: 10.1051/medsci/20092511885.L’organisation à grande échelle du chromosome bactérien Romain Mercier, Olivier Espéli, and Frédéric Boccard* Centre de génétique moléculaire du CNRS, 1, avenue de la Terrasse, 91198, Gif-sur-Yvette, France MeSH keywords: Ségrégation des chromosomes, Chromosomes de bactérie, Escherichia coli |
La grande taille des chromosomes comparée à la dimension des cellules impose une compaction des molécules d’ADN. Ainsi, le facteur de compaction du génome de cellules eucaryotes est de l’ordre de 105 et celui de cellules procaryotes de l’ordre de 103 [
1]. Cette compaction doit néanmoins permettre le bon déroulement des processus métaboliques associés à la molécule d’ADN (l’expression des gènes, la réplication de l’ADN, la ségrégation des chromosomes, le contrôle topologique de l’ADN). Cette compaction doit donc impliquer une organisation structurale des chromosomes précise et modulable. Un des éléments les mieux connus de l’organisation de la molécule d’ADN est le nucléosome qui enroule 140pb autour d’un octamère d’histone dans les cellules eucaryotes (voir revue récente dans Médecine/Sciences [
6]). Toutefois à plus grande échelle (> 1kb), les connaissances sont moins précises. Les études menées chez les bactéries ont montré que l’organisation de leur chromosome circulaire s’effectue à deux niveaux différents : (1) au niveau moléculaire, le surenroulement de l’hélice d’ADN génère la formation de microdomaines d’ADN surenroulé dont la taille est de l’ordre de 10 kb ; (2) au niveau cellulaire, le chromosome est organisé en 4 régions structurées, appelées macrodomaines (MD), d’une taille proche de 1Mb auxquelles s’ajoutent deux régions non structurées (régions NS) [
2,
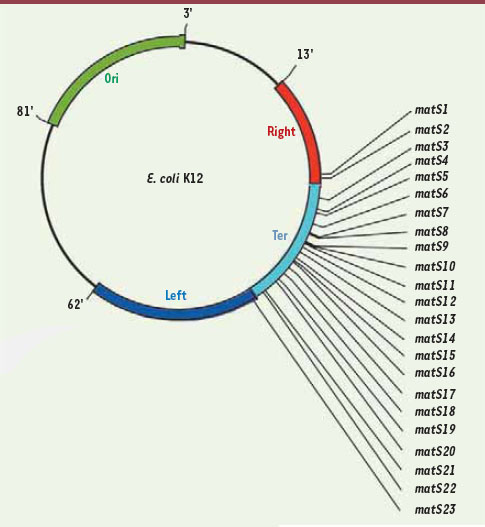
3]. La distinction MD/région NS a été révélée par un test génétique d’interaction à longue distance entre des séquences d’ADN [3]. Dans un macrodomaine, les collisions entre séquences se font à haute fréquence alors que deux séquences de deux macrodomaines différents n’interagissent pas. Les séquences des régions NS interagissent avec les séquences des MD adjacents. Le domaine Ori incluant l’origine de réplication oriC est flanqué des deux régions non structurées. Le domaine Ter comprenant la zone où se termine la réplication se situe entre les domaines Right et Left (Figure 1).
 | Figure 1.
Organisation du chromosome d’Escherichia coli en 4 macrodomaines et 2 régions non-structurées. Les macrodomaines et les régions non-structurées sont représentés sur la carte génétique du chromosome circulaire d’Escherichia coli (coordonnées en minutes (0-100’)). Les sites matS sont indiqués dans le macrodomaine Ter (de 27’ à 40’). |
|
Les macrodomaines et la dynamique du chromosome bactérien Au cours du cycle cellulaire, le chromosome doit être répliqué puis ségrégé dans les cellules filles. Alors que ces deux étapes du cycle cellulaire sont distinctes au niveau spatio-temporel dans les cellules eucaryotes, elles peuvent être concomitantes chez les bactéries. L’organisation du chromosome bactérien doit donc prendre en compte la dynamique du chromosome au cours de la ségrégation. Nos travaux de localisation des différentes régions du chromosome par microscopie à fluorescence avaient révélé l’influence des macrodomaines dans la dynamique du chromosome [
4]. Nous avons pu caractériser trois propriétés dans les MD qui les différencient des régions NS. L’ADN des chromosomes est très peu mobile dans la cellule, il diffuse dans un espace restreint de manière sub-diffusive. Nous avons fait les observations suivantes : d’une part les séquences contenues dans un macrodomaine ont une mobilité particulièrement faible dans la cellule alors que les séquences des régions NS ont une mobilité plus grande ; d’autre part, il y a au niveau des MD une étape de colocalisation des chromatides sœurs après réplication alors que cette étape n’est pas détectée dans les régions NS. Finalement, après ségrégation, les séquences contenues dans les MD vont chacune se concentrer dans un espace précis de la cellule alors que les séquences des régions NS ont un positionnement moins bien défini. Les MD isolent les chromosomes en quatre grands territoires chromosomiques dans la cellule. Cette organisation est assimilable à celle des territoires chromosomiques observés en interphase dans les cellules eucaryotes. Les raisons de cette organisation et les mécanismes responsables restaient inconnus. |
Organisation du macrodomaine Ter La compréhension des mécanismes de structuration du chromosome implique l’identification des déterminants des MD. Nous avions proposé que la structuration d’un MD pouvait reposer sur la reconnaissance par un facteur protéique de séquences multiples spécifiques d’un MD [3]. Pour tester cette hypothèse, nous avons recherché en collaboration avec Marie-Agnès Petit, Meriem El-Karoui, Sophie Schbath (INRA, Jouy-en-Josas) et Stéphane Robin (INRA, Paris) des motifs nucléotidiques répétés sur le chromosome et spécifiques d’un macrodomaine. Nous avons identifié un motif de 13 nucléotides (appelé matS pour macrodomain Ter site) présent en 23 exemplaires dans le macrodomaine Ter et absent du reste du chromosome (Figure 1) [
5]. La densité de sites matS dans cette région est d’environ 1 site tous les 35 kb. Par des approches de biochimie et de génétique, nous avons identifié et caractérisé un facteur protéique interagissant spécifiquement avec le motif matS (appelé MatP pour macrodomain Ter protein). Le motif matS et la protéine MatP sont conservés chez les entérobactéries ainsi que chez d’autres bactéries plus éloignées phylogénétiquement suggérant que l’organisation des chromosomes en macrodomaines pourrait être un mécanisme général conservé de structuration des chromosomes. |
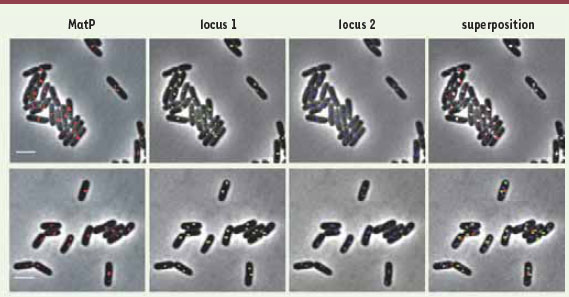
MatP confère ses propriétés au MD Ter Les analyses d’interaction de la protéine MatP avec le chromosome d’E coli montre que MatP reconnaît spécifiquement les séquences du macrodomaine Ter (Figure 2). Des expériences d’immunoprécipitation de chromatine ont montré de manière directe la liaison in vivo de la protéine MatP avec la majorité des sites matS. L’effet de la délétion du gène matP sur les propriétés du macrodomaine Ter révèle que MatP est le principal facteur protéique responsable de la structuration du macrodomaine Ter [5]. En effet en l’absence de MatP, le macrodomaine Ter présente des propriétés caractéristiques de région NS : (1) les séquences contenues dans le macrodomaine Ter peuvent interagir avec les séquences des macrodomaines adjacents, (2) la mobilité des séquences de la région Ter est identique à celle des régions NS, (3) l’étape de colocalisation des marqueurs après réplication est réduite, (4) le territoire chromosomique occupé par la région Ter est moins bien défini.  | Figure 2.
La protéine MatP définit un espace cellulaire occupé par le macrodomaine Ter. À gauche, le foyer formé par la protéine de fusion fluorescente MatP-mCherry (en rouge) se colocalise avec des marqueurs chromosomiques du macrodomaine Ter (indiqués en vert et en bleu). La colocalisation est révélée par la présence de foyers de couleur jaune, blanche ou magenta. À droite, le foyer formé par la protéine de fusion fluorescente MatP-mCherry (en rouge) est distinct des foyers de marqueurs localisés dans les macrodomaines Left et Right (indiqués en vert et bleu). |
|
MatP contrôle la ségrégation du chromosome Les propriétés conférées par la protéine MatP sont essentielles au bon déroulement du cycle cellulaire. Dans une souche mutante pour le gène matP, on observe un défaut de viabilité qui s’accompagne de la formation de deux types de cellules aberrantes : (1) de longues cellules filamenteuses qui résultent d’une incapacité à se diviser et qui accumulent un grand nombre de chromosomes non ségrégés, (2) des cellules anucléées ayant perdu leur chromosome [5]. Ces deux types cellulaires indiquent un défaut de ségrégation des chromosomes au cours du cycle cellulaire. Ces résultats mettent en lumière un rôle de l’organisation du macrodomaine Ter dans la ségrégation du chromosome. L’identification des déterminants de la structuration d’un MD ouvre la voie pour la caractérisation moléculaire de mécanismes responsables de la prise en charge d’une grande région chromosomique. Ces résultats permettront également la recherche de partenaires de MatP impliqués dans la chorégraphie du chromosome au cours du cycle cellulaire. Ils révèleront ainsi les structures qui assurent au chromosome son positionnement dynamique dans la cellule.

|
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Holmes VF, Cozzarelli NR. Closing the ring: links between SMC proteins and chromosome partitioning, condensation, and supercoiling. Proc Natl Acad Sci USA 2000; 97 : 1322–4. 2. Niki H, Yamaichi Y, Hiraga S. Dynamic organization of chromosomal DNA in Escherichia coli. Genes Dev 2000; 14 : 212–23. 3. Valens M, Penaud S, Rossignol M, Cornet F, Boccard F. Macrodomain organization of the Escherichia coli chromosome. EMBO J 2004; 23 : 4330–41. 4. Espeli O, Mercier R, Boccard F. DNA dynamics vary according to macrodomain topography in the E. coli chromosome. Mol Microbiol 2008; 68 : 1418–27. 5. Mercier R, Petit MA, Schbath S, et al. The MatP/matS site-specific system organizes the terminus region of the E. coli chromosome into a macrodomain. Cell 2008; 135 : 475–85. 6. Bertin A, Mangenot S. Structure et dynamique de la particule cœur de nucléosome. Med Sci (Paris) 2008; 24 : 715–9. |